O projeto CiteULike.
O projeto consiste em estudos realizados pelo Grupo de Sistemas Complexos do Instituto de Física da UFF, da rede de compartilhamento de artigos CiteULike, que agrupa para cada perfil (usuário) um conjunto de artigos que são selecionados pelo utilizador do perfil nas mais diversas revistas de fomentação cientifica. Esses artigos são caracterizado por tags, palavras chaves que indicam o assunto a qual o artigo está relacionado. Uma base de dados é disponibilizada pelo próprio site, que fornece informações de um período desde 2004 até os dias de hoje, contendo todas as informações das evolução e conexão dos perfis tais como: artigos, tags e evolução temporal do crescimento de cada perfil. Várias perguntas podem ser feitas acerca dessa estrutura, perguntas que nos remete a um estudo que relaciona as redes complexas bem como um estudo sobre a correlação de grupos de interesses ou comunidades de assuntos e interesses comuns a partir da análise dessas tags. Inicialmente, varreremos toda base de dados em buscas de estatísticas macro, tais como os mil artigos mais importantes, para que sob um número menor de amostras, possamos melhorar a precisão e ganho de tempo computacional.
Descrição do Banco de Dados. A base de dados fornecida pelo CiteULike é estruturada da seguinte forma: Coluna 1- (identificação dos usuários)
Coluna 2 – (identificação dos artigos) Coluna 3- (Evolução temporal) Coluna 4 – (tags dos artigos)
A estrutura se constrói considerando que a usuário se repete para cada nova tag adiciona a um artigos, de maneira que que se um artigo tem 10 tags, o usuário se repetirá 10 vezes juntamente com o artigo.
| Usuários | Artigos | Evolução | Tags |
|---|
| usuário1 | artigo_a | (####) | tag_a1 |
| usuário1 | artigo_a | (####) | tag_a2 |
| usuário1 | artigo_a | (####) | tag_a3 |
| usuário1 | artigo_a | (####) | tag_a4 |
| usuário1 | artigo_a | (####) | tag_a5 |
| usuário1 | artigo_b | (####) | tag_b1 |
| usuário1 | artigo_b | (####) | tag_b2 |
| usuário1 | artigo_b | (####) | tag_b3 |
| … | … | … | … |
Na tabela acima, o usuário 1 possui 2 artigos e 8 tags em seu perfil. Nossa proposta inicial é filtrar toda a base de dados, que compreende um período de evolução do CiteULike de 2004 a data atual com aproximadamente 10.000.000 linhas. Serão filtrados os 1000 artigos mis citados. Iremos considerar em nossa análise apenas a segunda e quarta coluna, uma vez que é nosso interesse montar uma rede entre os artigos conectada por tags comuns.
O código que filtra os nmax artigos mais citados é:
# Abrindo arquivo de dado separados por "|" open FILE, "data.dat" or die $!; open ORDEM, ">ordem.dat" ; $i=0; # Criando listas vazias @cont=(); # @ncont=(); @tags_artigos=(); while(<FILE>) { chomp; $i++; @lista= split(/\|/,$_); $artigos[$i]=$lista[0]; $usuarios[$i]=$lista[1]; $tags[$i]=$lista[3]; # Acumula a frequencia de artigos @cont{$lista[0]}++; # Escrevendo as tags de cada artigo $ncont{$lista[0]}++; $tags_artigos{$ncont{$lista[0]}}{$lista[0]}=$lista[3]; # Guarda as tags de cada artigo } #Nota: O caractere "|" é especial e seu uso deve ser precedido de "\". Orácio me passou esse detalhe. close FILE; $n=$i; # Número de linhas da base de dados $nmax=300; # nmax artigos mais citados # Ordena os artigos por ordem de citações @ordem = sort { $cont{$b} <=> $cont{$a} } keys %cont; for ($i=0;$i<=$nmax;$i++) { # Escreve um arquivo de dados com os nmax artigos mais citados for ($j=1;$j<=$ncont{$ordem[$i]};$j++) { print ORDEM "$ordem[$i] $tags_artigos{$j}{$ordem[$i]} \n"; } }
Esse código retorna um arquivo “ordem.dat” que organiza em ordem decrescente os nmax artigos mais citado. Varremos toda a base de dados (11.000.000 de linhas) e retornamos em “ordem.dat” os 1.000 artigos mais citados. O arquivo segue a mesma etrutura da tabela citada anteriormente, no entanto são armazenados apenas os artigos e suas respectivas tags. Click aqui (ordem.dat.tar.gz)para fazer o download do arquivo.
Gerando rede entre artigos mediante um parâmetro “w”.
Seguindo a estrutura do arquivo “ordem.dat”,para cada artigo exitem um número de tags “n” que caracteriza seu conteúdo. Cada artigo será representado por um vetor e cada domínio será ocupado por uma tag.
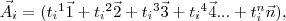
onde 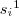 é a primeira tag do artigo
é a primeira tag do artigo  que pertence a “direção”
que pertence a “direção” 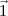 , aqui definida.
Dessa forma, o artigo
, aqui definida.
Dessa forma, o artigo 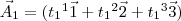 possui as tags
possui as tags 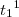 ,
, 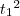 e
e 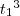 nas respectivas “direções”:
nas respectivas “direções”: 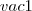
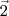 e
e 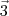 .
Para facilitar a notação, rescreveremos a representação do mesmo artigo da seguinte forma
.
Para facilitar a notação, rescreveremos a representação do mesmo artigo da seguinte forma
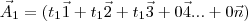
Para um número nmax de artigos, iremos gerar uma rede que tem como parâmetro de ligação a interseçaõ entre as tags de cada artigo, de modo que pode ser definida entre dois artigos qualquer pertencentes a rede, a distância interpretada por
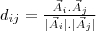
O parãmetro w é a varoável de corte. Se <latex>⇐<\latex>, a conexão entre os artigos <latex><\latex> e <latex><\latex> é estabelecida.
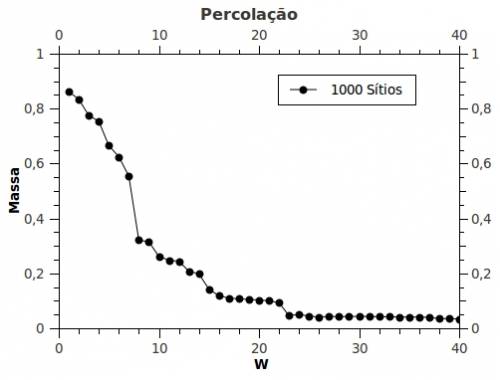
Para um conjunto de artigos estabelecemos uma variação do parâmetro de corte w. Para cada estrutura formada, calculamos o tamanho do maior cluster (cluster percolante).
Foram usados dois scriptes em Perl. O primeiro para organizar a estrutura da rede mãe. O segundo para fazer a estatística com a variação de w.
Estrutura da rede
open FILE, "ordem2.dat" or die $!; #open ARRAYS, ">arrays.dat"; #open CONEX, ">conex.dat"; open FREDE, ">frede2.dat"; open NET, ">rede2.net"; open REDE, ">rede2.dat"; $w=1; $i=0; while (<FILE>) { chomp; @lista= split(" ",$_); $i++; $artigos[$i]=$lista[0]; $tags[$i]=$lista[1]; } close FILE; @cont=(); @idtags=(); for($ii=1;$ii<=$i;$ii++) { $cont{$artigos[$ii]}++; $idtags{$artigos[$ii]}{$cont{$artigos[$ii]}}=$tags[$ii]; } @id=keys(%cont); $d=$#id+1; print "$d \n"; # Escrevendo as Tags de cada Artigo #for($i=1;$i<=$#id+1;$i++) #{ #print ARRAYS "$id[$i] '-------------------' \n"; #for($j=1;$j<=$cont{$id[$i]};$j++) #{ #print ARRAYS "$idtags{$id[$i]}{$j} \n"; #} #} #close ARRAY; # Formando rede por conexão de Tgas @nv=(); @vz=(); @peso=(); for($i=1;$i<=$d-1;$i++) { for($j=$i+1;$j<=$d;$j++) { #print CONEX "$id[$i] '------->' $id[$j] \n"; %ng=(); @conex=(); for($ii=1;$ii<=$cont{$id[$i]};$ii++) { for($jj=1;$jj<=$cont{$id[$j]};$jj++) { if($idtags{$id[$i]}{$ii} eq $idtags{$id[$j]}{$jj}) { $ng{$idtags{$id[$i]}{$ii}}=1; } } } @conex=keys(%ng); $l=$#conex+1; #print CONEX "'------------------------------' \n"; #print CONEX "@conex \n"; #print CONEX "$l \n"; if($l>=$w) { $nv[$i]++; $nv[$j]++; $vz{$i}{$nv[$i]}=$j; $vz{$j}{$nv[$j]}=$i; $peso{$i}{$j}=$l } } } # Gerando Histogramas @fl=(); print NET "*Network \n"; print NET "*Vertices $#id \n"; for($i=1;$i<=$d;$i++) { print NET "$i '$id[$i]' \n"; $fl{$nv[$i]}++; } for($i=1;$i<=$d;$i++) { if($fl{$i}>0) { print FREDE "$i $fl{$i} \n"; } } close FREDE; print NET "*Edges \n"; for($i=1;$i<=$d;$i++) { for($j=1;$j<=$nv[$i];$j++) { #if($peso{$i}{$vz{$i}{$j}}>1) #{ print NET "$i $vz{$i}{$j} 1 \n";#$peso{$i}{$vz{$i}{$j}} \n"; print REDE "$i $vz{$i}{$j} $peso{$i}{$vz{$i}{$j}} \n"; #} } }
Calculo do maior cluster
open GRAFICO, ">grafico.dat"; open CLUSTERS, ">clusters.dat"; for($w=1;$w<=50; $w++) { open FILE, "rede.dat" or die $!; %vz=(); %nv=(); @nv=(); @vz=(); @peso=(); %peso=(); @lista=(); %lista=(); while (<FILE>) { chomp; @lista= split(/ /,$_); if($lista[2]>=$w) { $nv{$lista[0]}++; # $nv{$lista[1]}++; $vz{$lista[0]}{$nv{$lista[0]}}=$lista[1]; # $vz{$lista[1]}{$nv{$lista[1]}}=$lista[1]; $peso{$lista[0]}{$lista[1]}=$lista[2]; } } $pp=0; $l=0; @id=(); %id=(); @id=keys(%nv); #@id=sort {$a <=> $b}(@id); $l=$#id+1; $id[$l]=$id[0]; $w2=500-$l; #Calculando o Cluster Percolante @acc=(); %acc=(); $nn=0; @si=(); %si=(); $ni=0; %pai=(); for($i=1;$i<=$l;$i++) { $pai{$id[$i]}=0; $si[$id[$i]]=0; $acc[$id[$i]]=0; } for ($h=1;$h<=$l;$h++) { if($pai{$id[$h]} eq 0) { $pp++; $nn=$nv{$id[$h]}; $pai{$id[$h]}=$pp; $acc[$pp]++; $ni=$nn; for($r=1;$r<=$ni;$r++) { $si[$r]=$vz{$id[$h]}{$r}; } while($nn ne 0) { $nn=0; for($i=1;$i<=$ni;$i++) { for($j=1;$j<=$nv{$si[$i]};$j++) { if($pai{$vz{$si[$i]}{$j}} eq 0) { $pai{$vz{$si[$i]}{$j}}=$pp; $acc[$pp]++; $nn++; $si[$nn]=$vz{$si[$i]}{$j}; } } $ni=$nn; } } } } ##### Fim do algorítimo for($u=1;$u<=$pp;$u++) { print CLUSTERS " $u '------------->' $acc[$u] \n"; for($i=1;$i<=$l;$i++) { if($pai{$id[$i]} eq $u) { print CLUSTERS "$id[$i] \n"; } } } my $max=1; for($i=1;$i<=$pp;$i++) { #print "$acc[$i] \n"; if($acc[$i]>=$max) { $max=$acc[$i]; } } $s=($pp+$w2)/500; $s1=$w/1000; $s2=$max/1000; $s3=$pp/$l; print GRAFICO "$w $s2 \n"; print "$w $s2 \n"; print CLUSTERS " ################ \n"; }